Перейти к:
Глубокое обучение в выявлении раннего папиллярного рака мочевого пузыря на ограниченном наборе цистоскопических изображений
https://doi.org/10.47093/2218-7332.2024.953.15
Аннотация
Цель. Разработать и изучить эффективность сверточной нейронной сети (СНС) в обнаружении папиллярного рака мочевого пузыря (РМП) на ограниченном наборе цистоскопических изображений.
Материалы и методы. В исследование включены 20 пациентов, которым проведена цистоскопия в белом свете и гистологически подтвержден папиллярный рак мочевого пузыря. Набор данных состоял из 125 изображений, извлеченных и размеченных врачом-урологом: 88 изображений – папиллярные опухоли, 37 – здоровая ткань стенки мочевого пузыря. Для обучения были отобраны 100 изображений, а для тестирования – 25. Использована архитектура U-net и модель СНС VGG16. Для каждого кадра вручную создавалась двоичная маска на основе комментариев, предоставленных врачом-урологом. Каждое изображение было дополнительно обработано для совместимости с моделью, на вход подавались изображения размером 224×224 пикселя для уменьшения количества параметров. С целью расширения набора данных применили вертикальные и горизонтальные повороты в сочетании со случайными вращениями. Рассчитана метрика Дайса, чувствительность, специфичность, доля ложноположительных и ложноотрицательных значений, точность; площадь под ROC-кривой.
Результаты. Для исходного набора данных получены следующие показатели: специфичность 84,56%, чувствительность 82,18%, доля ложноположительных результатов 15,44%, ложноотрицательных 17,82%, точность 76,40%, метрика Дайса – 83,16%. Для расширенного набора данных получены следующие показатели: специфичность 82,99%, чувствительность 82,70%, доля ложноположительных результатов 17,01%, ложноотрицательных 17,30%, точность 74,72%, метрика Дайса 82,82%. Площадь под ROC-кривыми составила 92,93% для исходного набора данных и 91,69% для расширенного набора данных.
Заключение. Признаки раннего папиллярного РМП при анализе цистоскопических изображений можно обнаружить с помощью созданной СНС. Результаты исследования могут служить отправной точкой для развития новых методов диагностики РМП с использованием технологий глубокого обучения.
Ключевые слова
Список сокращений:
- ИИ – искусственный интеллект
- РМП – рак мочевого пузяря
- СНС – сверточная нейронная сеть
Рак мочевого пузыря (РМП) занимает 10-е место в мировой статистике злокачественных опухолей: ежегодно диагностируется примерно 550 000 новых случаев и почти 200 000 смертей, в ближайшее время ожидается рост этих показателей [1][2].
Основным методом диагностики РМП является цистоскопия, во время которой эндоскоп через уретру вводится в мочевой пузырь с целью визуального осмотра его внутренней оболочки. При обнаружении любых подозрительных образований может быть проведена трансуретральная резекция для забора ткани на биопсию с целью оценки типа, подтипа и класса опухоли. Более того, трансуретральная резекция является первой линией лечения РМП, используемой для иссечения уротелиальных опухолей, и часто выполняется в момент установления диагноза. Однако назначение трансуретральной резекции зависит от результатов визуального осмотра, который является субъективным и подвержен ошибкам в зависимости от опыта оператора.
Действительно, цистоскопия в белом свете, являющаяся в настоящее время наиболее распространенным методом диагностики РМП, имеет частоту пропусков ранних стадий немышечных инвазивных папиллярных опухолей (стадии Ta и T1) до 24,9% по сравнению с фотодинамической цистоскопией [3]. Несмотря на разработку более надежных методов, таких как фотодинамическая и узкополосная визуализация [4], цистоскопия в белом свете остается одной из стандартных диагностических процедур для РМП3.
Автоматизированное обучение – это быстро развивающаяся область исследований в медицинской визуализации и радиологии, в которой используется искусственный интеллект (ИИ) с целью минимизации ошибок и облегчения интерпретации и принятия решений [5][6]. Системы автоматизированного проектирования с применением глубокого обучения разрабатываются для тщательного анализа изображений в различных областях медицины [7]. Все больше фактов демонстрируют возможность применения глубокого обучения в обработке изображений магнитно-резонансной и компьютерной томографии [8–11], высокой точности обнаружения полипов при колоноскопии [12][13], диагностике РМП по данным цистоскопии [14].
Модели глубокого обучения обычно требуют очень больших наборов данных для обучения, состоящих из сотен тысяч изображений. Во время каждой эпохи весь набор данных просматривается, что позволяет алгоритму эффективно регулировать веса и одновременно обучаться на крайне неоднородных выборках. Но получение таких объемов данных с учетом того, что каждое изображение прокомментировано клиническим специалистом, не представляется возможным в контексте получения медицинских изображений. Чтобы преодолеть это ограничение, можно использовать метод, известный как расширение набора данных, для искусственного дополнения имеющегося набора данных [15]. Использование расширения данных позволяет создавать случайные изменения для каждой эпохи вместо многократного обучения на одном и том же исходном наборе данных. Эти изменения должны быть выбраны с учетом специфики проблемы на основе образцов, присутствующих в исходном наборе данных.
Цель данного исследования – разработать и изучить эффективность сверточной нейронной сети (СНС) в обнаружении РМП и сегментации папиллярного рака на ограниченном наборе цистоскопических изображений.
МАТЕРИАЛЫ И МЕТОДЫ
Сбор данных
Видеоматериалы для данного исследования были получены во время цистоскопических исследований, проведенных в УКБ № 2 Первого МГМУ им. И.М. Сеченова врачами-урологами по стандартной методике. В исследование включены 20 последовательных пациентов, набранных после начала исследования. Критерием включения служили гистологически подтвержденный диагноз «папиллярный РМП» и наличие записи цистоскопии.
Информативные изображения были извлечены и размечены врачом-урологом Лекаревым В.Ю. Окончательный набор данных состоял из 125 изображений, из которых 88 изображений – папиллярные опухоли, а 37 изображений демонстрировали здоровую ткань стенки мочевого пузыря.
Подготовка набора данных
Двоичные маски были созданы для каждого кадра на основе комментариев, предоставленных медицинским экспертом. Поскольку цистоскопические изображения обычно круглые, двоичные маски подгонялись вручную, чтобы исключить темные участки вокруг. Мы присваивали пикселю значение 1, если он попадал в границу опухолевой области, и 0 – в противном случае. К обучающей выборке отнесены 80% данных, а остальные 20% использовались для окончательного тестирования модели. Во избежание переобучения модели изображения, полученные у одного и того же пациента, включались либо в обучающую, либо в тестовую выборку. В результате обучающий набор содержал 100 изображений и соответствующих им масок для сегментации. Окончательный тестовый набор содержал 25 изображений и соответствующие им сегментационные маски.
Архитектура глубокого обучения
Модель глубокого обучения была реализована на Python с использованием библиотек Keras и Tensorflow. Для определения контуров опухолей использовалась СНС, в частности конструкция кодер-декодер. Для кодирующей части была использована архитектура VGG16 [16] и заданы веса из модели VGG16, предварительно обученной на наборе данных ImageNet [17]. Декодер был разработан в соответствии с принципами архитектуры UNet [18] и обучен с нуля. Модель получала изображение размером 224×224 пикселей и создавала прогнозируемые маски сегментации того же размера, что и входные данные. Всего было 24 118 502 параметра, из которых все, кроме 434, являлись обучаемыми.
Обработка данных
Все изображения требовали предварительной обработки для совместимости с используемой моделью. Исходные изображения и соответствующие маски с пояснениями были уменьшены до размера 224×224 пикселей, чтобы уменьшить количество параметров в СНС. Кроме того, необработанные RGB-изображения были выровнены по центру путем вычитания средних значений по каждому каналу, рассчитанных на основе набора данных ImageNet.
Обучение модели и оценка производительности
Модель обучалась с помощью функции потерь и оценивалась на тестовом наборе после каждой эпохи. Для определения оптимального количества эпох, необходимых для обучения, мы использовали метод «локтя», т.е. прекращали обучение, как только потери при проверке достигали своего глобального минимума и начинали расти: это означало, что модель чрезмерно подстраивается.
Модель глубокого обучения
Для обнаружения границ опухоли на цистоскопических изображениях мы подобрали и внедрили СНС, разработанную специально для задач сегментации изображений. Архитектура нашей системы глубокого обучения состояла из двух фаз: кодирования и декодирования, схематично показанных на рисунке 1. Такая структура позволяет СНС извлекать из изображения сложные и абстрактные признаки, а затем восстанавливать пространственные отношения между ними. Перед анализом все изображения были изменены в размерах и выровнены для обеспечения совместимости с СНС. Модель получала исходное цистоскопическое изображение и выдавала двоичную маску, где 1 присваивалась пикселям в пределах предсказанной области опухоли, а 0 – в противном случае. Мы подготавливали нашу модель на обучающем наборе данных, минимизируя потери, опираясь на метрику Дайса [19], которая обычно используется в задачах сегментации изображений, поскольку она может эффективно работать с сильно несбалансированными классами. Модель оценивалась после каждой эпохи с использованием подтвержденного набора данных, чтобы предотвратить переобучение.
Модель состоит из двух этапов: кодирования и декодирования. Кодер получает на вход исходное цистоскопическое изображение и извлекает представления низшего и высшего уровней. После каждого блока сверток применяется операция подвыборки для создания схем признаков низкого разрешения. Эти схемы затем поступают в декодер для восстановления пространственной информации и создания высокоразмерной маски сегментации. Белые пиксели в бинарной маске определяют прогнозируемую область опухоли, черные пиксели – окружающую здоровую ткань (рис. 1).
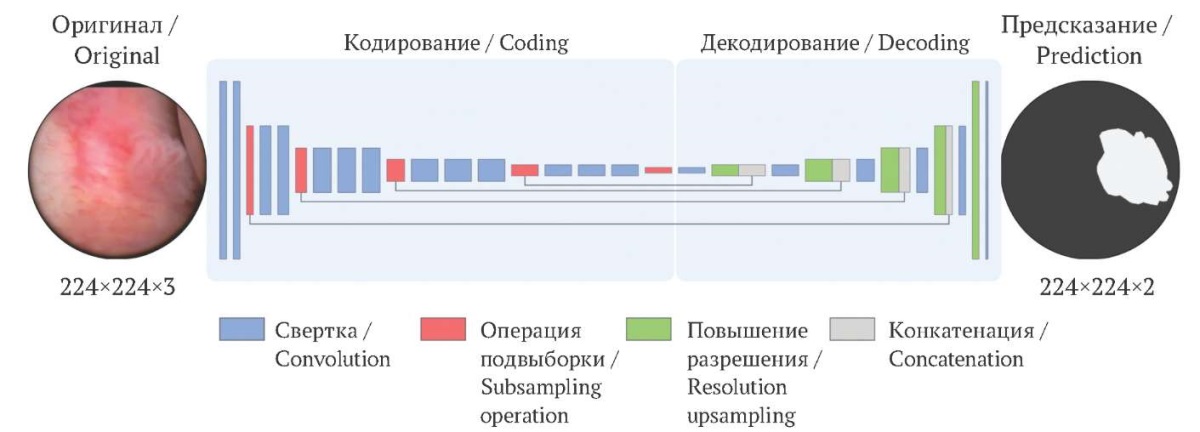
РИС. 1. Схематическая иллюстрация архитектуры сверточной нейронной сети.
FIG. 1. Schematic illustration of the convolutional neural network architecture.
Расширение данных
Для расширения набора данных с учетом специфики изучаемой проблемы – папиллярная опухоль может выступать из любой части мочевого пузыря и наблюдаться под разными углами – мы применили вертикальные и горизонтальные повороты в сочетании со случайными вращениями, как показано на рисунке 2.
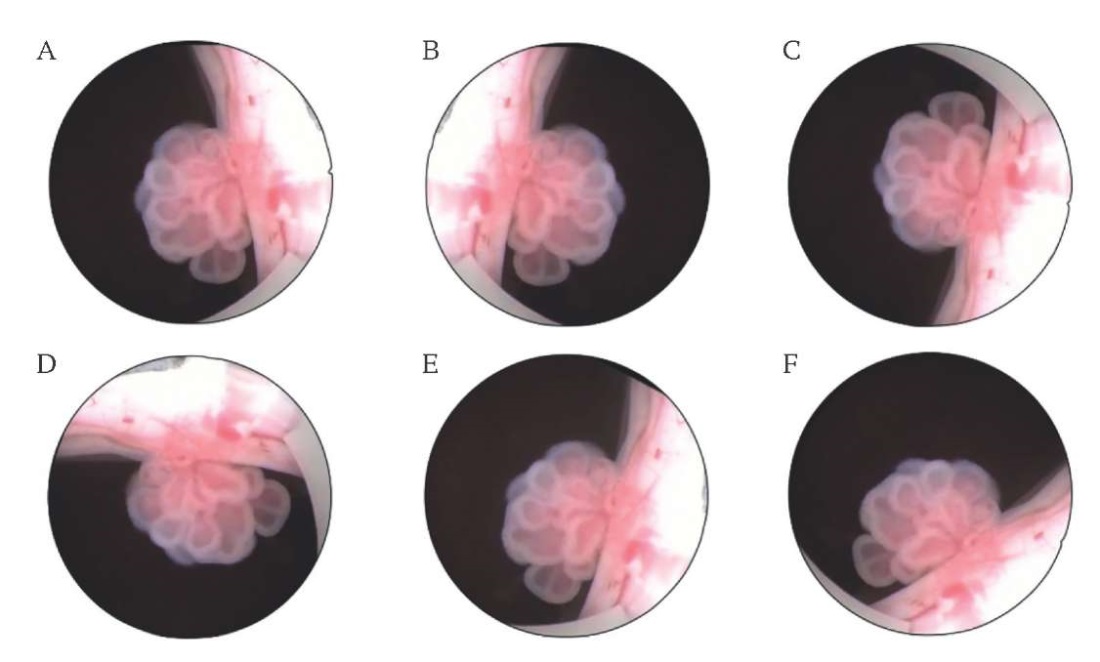
РИС. 2. Увеличение набора данных за счет вращения исходного изображения.
A. Исходное изображение.
B. Горизонтальный поворот.
C. Вертикальный поворот.
D–F. Случайное вращение исходного изображения.
FIG. 2. Augmenting the dataset by rotating the original image.
A. Original image.
B. Horizontal rotation.
C. Vertical rotation.
D–F. Random rotations of the original image.
Статистический анализ
Эффективность итоговой модели оценивалась на тестовом наборе с помощью ряда метрик, включая метрику Дайса, чувствительность, специфичность, долю ложноположительных и ложноотрицательных значений, точность; построены ROC-кривые и рассчитана площадь под ROC-кривой.
РЕЗУЛЬТАТЫ
Сравнение производительности модели, обученной на исходном наборе данных и на расширенном наборе, представлено в таблице.
Таблица. Показатели производительности моделей,
обученных на исходном и на расширенном наборах данных
Table. Performance metrics of models
trained on the original and augmented dataset
|
Характеристики / Measures |
Исходный набор данных / Original dataset |
Расширенный набор данных / Augmented dataset |
|
Специфичность / Specificity, % |
84,56 |
82,99 |
|
Чувствительность / Sensitivity, % |
82,18 |
82,70 |
|
Доля ложноположительных значений / False positive rate, % |
15,44 |
17,01 |
|
Доля ложноотрицательных значений / False negative rate, % |
17,82 |
17,30 |
|
Точность / Accuracy, % |
76,40 |
74,72 |
|
Метрика Дайса / Dice coefficient, % |
83,16 |
82,82 |
Обучение модели на расширенном наборе данных привело к снижению частоты ложноотрицательных показателей с 17,82 до 17,30% (табл.).
Как видно из ROC-кривых, представленных на рисунке 3, модель, обученная на оригинальных изображениях, работает немного лучше по сравнению с подходом, основанном на расширении данных. Площадь под ROC-кривыми была равна 92,93 и 91,69% соответственно.
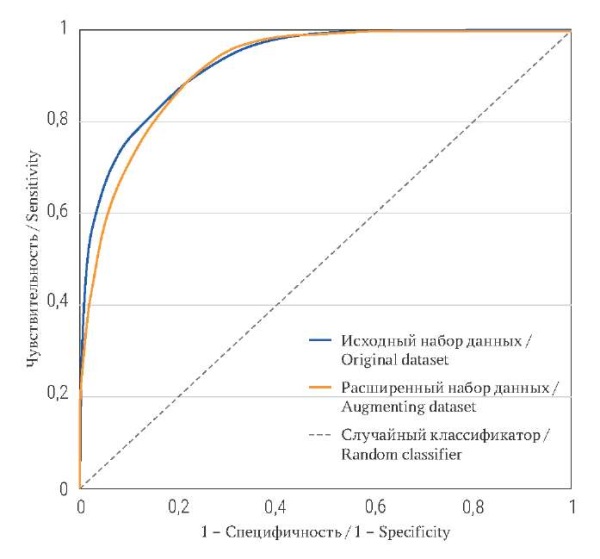
РИC. 3. ROC-кривые для моделей, обученных на исходном и расширенном наборах данных.
FIG. 3. ROC curves trained on the original and augmented dataset.
Модель показала хорошие результаты. Это видно из результатов анализа модели и при сравнении их с комментариями экспертов, полученными на основе данных (рис. 4). Согласно полученным результатам, модель способна распознавать характерные малозаметные признаки злокачественных образований, что приводит к точному определению границ опухоли.

РИС. 4. Примеры обнаружения опухолей и сегментации поражений
на цистоскопических изображениях из тестового набора.
FIG. 4. Examples of tumor detection and lesion segmentation
on cystoscopic images from the test set.
Примечание: СНС – сверточная нейронная сеть;
розовый цвет – неизмененная слизистая; серо-голубой – опухоль.
Note: CNN – convolutional neural network;
pink color – unchanged mucosa; gray-blue – tumor.
ОБСУЖДЕНИЕ
Последние достижения в области машинного обучения способствуют технологическому прогрессу в различных областях. В здравоохранении особый интерес в последние годы вызывает компьютерная интерпретация радиологических и патологических данных, которая позволяет снизить рабочую нагрузку, давая врачам возможность сосредоточиться на более важных задачах. Очевидное преимущество можно заметить в передаче повторяющейся, умственно сложной и утомительной работы машинам, которые способны обеспечить более быструю и точную интерпретацию, повышая производительность и минимизируя контроль.
Направление ИИ под названием «глубокое обучение», в частности с использованием СНС, нашло свое применение в анализе различных данных медицинской визуализации. В зависимости от архитектуры СНС может быть выполнен ряд задач, включая классификацию заболеваний, распознавание поражений, сегментацию тканей и описание органов. Таким образом, глубокое обучение обеспечивает гибкость в обработке изображений в зависимости от конкретных клинических целей диагностики.
Пожалуй, самые яркие примеры диагностики с использованием глубокого обучения можно увидеть в радиологии. К ним относятся: выявление и стадирование рака легкого на основании данных компьютерной томографии [20], определение границ глиомы на предоперационных мультимодальных магнитно-резонансных томограммах [21], классификация образований при маммографических исследованиях [22][23]. Кроме того, глубокое обучение было успешно применено для выявления диабетической ретинопатии на фундус-фотографиях сетчатки [24] и для разделения кожных новообразований на злокачественные и доброкачественные [25]. Такие системы автоматизированного обучения потенциально могут способствовать улучшению результатов лечения онкологических пациентов за счет усовершенствования тестов для раннего выявления заболеваний и повышения эффективности диагностики [26].
В данном исследовании рассматривается применение глубокого обучения для обнаружения папиллярного РМП. Мы предложили использовать специализированную архитектуру СНС, предназначенную для сегментации цистоскопических изображений.
Одним из основных ограничивающих факторов, с которым мы столкнулись в нашем исследовании, характерном для медицинских исследований ИИ, был небольшой размер набора данных и отсутствие комментариев в других наборах данных. Мы предположили, что настройка СНС, ранее обученных на большем наборе данных, таком как ImageNet, может привести к более высокой производительности, чем обучение модели с нуля. Поэтому был использован заранее обученный VGG16, реализованный в качестве этапа кодирования модели.
Кроме того, исследование, посвященное обнаружению полипов на изображениях колоноскопии, показало, что различные стратегии расширения данных могут принести большую пользу при работе с ограниченным объемом данных [27]. Учитывая характер цистоскопического исследования, мы использовали вертикальные и горизонтальные перевороты, а также случайные повороты для имитации различных углов камеры. Мы исследовали, как такие изменения влияют на работу модели, и обнаружили, что, хотя общая оценка была немного хуже, удалось повысить чувствительность.
Данная работа иллюстрирует лишь первый шаг в долгом процессе внедрения системы автоматизированного обучения в клиническую практику. Необходимо провести проспективное клиническое исследование, чтобы оценить, приведет ли внедрение системы распознавания, дополненной ИИ, к увеличению чувствительности при обнаружении папиллярного РМП. В частности, можно ли обнаружить и удалить больше опухолей на ранних стадиях.
ЗАКЛЮЧЕНИЕ
Созданная в работе нейросетевая модель показывает, что алгоритмы глубокого обучения способны обнаруживать признаки раннего папиллярного РМП с точностью метрики Дайса 82,82% для расширенного набора данных. Подобная модель может стать помощником врача при анализе цистоскопических изображений. Для широкого применения глубокого обучения в клинической практике необходимы дальнейшие исследования, включая масштабные клинические испытания и оценку эффективности алгоритма в реальных условиях. Тем не менее результаты данного исследования могут служить отправной точкой для развития новых методов диагностики РМП с использованием технологий глубокого обучения.
ВКЛАД АВТОРОВ
В.C. Розова разрабатывала нейросетевую модель, анализировала результаты, участвовала в написании рукописи. К. Руссо разрабатывал нейросетевую модель, анализировал результаты. В.Ю. Лекарев и А.М. Дымов осуществляли подготовку диагностических исследований, разметку изображений, анализ результатов. В.В. Казанцева участвовала в подготовке графических материалов и написании рукописи. А.С. Ржевский руководил исследованием, участвовал в получении первичных результатов. А.В. Звягин руководил исследованием, участвовал в написании рукописи.
Все авторы одобрили окончательную версию статьи.
AUTHOR CONTIBUTIONS
Vlada S. Rozova developed artificial neural network-based model, analyzed the study results, and wrote the manuscript. Carlo Russo developed artificial neural network-based model, analyzed the study results. Vladimir Y. Lekarev and Alim M. Dymov prepared datasets, labeled medical images, and analyzed the results. Vlada V. Kazantseva preparade figures and wrote the manuscript. Alexey S. Rzhevskiy led the study and managed the primary data. Andrey V. Zvyagin led the study and wrote the manuscript.
All authors approved the final version of the article.
Соответствие принципам этики. Исследование проведено в соответствии с разрешением Локального этического комитета ФГБОУ ВО «Первый МГМУ им. И.М. Сеченова» Минздрава России (Сеченовский университет) № 22-21-09.12.2021. Все пациенты, включенные в исследование, дали письменное информированное согласие на обработку изображений цистоскопии. Все данные пациентов были обезличены перед дальнейшим изучением.
Доступ к данным исследования. Данные, подтверждающие выводы этого исследования, можно получить у авторов по обоснованному запросу.
Политика открытых данных: код для воспроизведения результатов доступен в открытом доступе: https://github.com/vladarozova/bladder-cancer/tree/master
Конфликт интересов. Авторы заявляют об отсутствии конфликта интересов.
Финансирование. Исследование проведено за счет гранта Российского научного фонда № 22-24-201291.
Ethics statements. The study was conducted in accordance with the permission of the Local Bioethics Ethical Committee of the Sechenov First Moscow State Medical University (Sechenov University), No. 22-21, of 09.12.2021. Written informed consent was obtained from all patients included in the study for the processing of cystoscopy images. The data of the patients were anonymized before the study.
Data availability. The data that support the findings of this study are available from the corresponding authors on reasonable request.
Data Availability Statement. The code for reproducing the results is openly available at https://github.com/vlada-rozova/bladder-cancer/tree/master.
Conflict of interests. The authors declare that there is no conflict of interests.
Financial support. The study was supported by the grant No. 22-24-201292 from the Russian Science Foundation.
1. https://rscf.ru/prjcard_int?22-24-20129&ysclid=lp6oqoxwl8139402339 (дата обращения: 16.11.2023).
2. https://rscf.ru/prjcard_int?22-24-20129&ysclid=lp6oqoxwl8139402339 (дата обращения: 16.11.2023).
3. Клинические рекомендации. Рак мочевого пузыря. https://cr.minzdrav.gov.ru/schema/11_3 (дата обращения: 17.08.2023).
Список литературы
1. Sung H., Feобоrlay J., Siegel R.L., et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 2021 May; 71(3): 209–249. https://doi.org/10.3322/caac.21660. Epub 2021 Feb 4. PMID: 33538338
2. van Hoogstraten L.M.C., Vrieling A., van der Heijden A.G., et al. Global trends in the epidemiology of bladder cancer: challenges for public health and clinical practice. Nat Rev Clin Oncol. 2023 May; 20(5): 287–304. https://doi.org/10.1038/s41571-023-00744-3. Epub 2023 Mar 13. PMID: 36914746
3. Burger M., Grossman H.B., Droller M., et al. Photodynamic diagnosis of non-muscle-invasive bladder cancer with hexaminolevulinate cystoscopy: a meta-analysis of detection and recurrence based on raw data. Eur Urol. 2013 Nov; 64(5): 846–854. https://doi.org/10.1016/j.eururo.2013.03.059. Epub 2013 Apr 8. PMID: 23602406
4. Tully K., Palisaar R.J., Brock M., et al. Transurethral resection of bladder tumours: established and new methods of tumour visualisation. Transl Androl Urol. 2019 Feb; 8(1): 25–33. https://doi.org/10.21037/tau.2018.12.12. PMID: 30976565; PMCID: PMC6414343
5. Fogel A.L., Kvedar J.C. Artificial intelligence powers digital medicine. NPJ Digit Med. 2018 Mar 14; 1: 5. https://doi.org/10.1038/s41746-017-0012-2. PMID: 31304291; PMCID: PMC6548340
6. Haug C.J., Drazen J.M. Artificial intelligence and machine learning in clinical medicine, 2023. N Engl J Med. 2023 Mar 30; 388(13): 1201–1208. https://doi.org/10.1056/NEJMra2302038. PMID: 36988595
7. Egger J., Gsaxner C., Pepe A., et al. Medical deep learning-A systematic meta-review. Comput Methods Programs Biomed. 2022 Jun; 221: 106874. https://doi.org/10.1016/j.cmpb.2022.106874. Epub 2022 May 11. PMID: 35588660
8. Wu Z., Wang F., Cao W., et al. Lung cancer risk prediction models based on pulmonary nodules: A systematic review. Thorac Cancer. 2022 Mar; 13(5): 664–677. https://doi.org/10.1111/1759-7714.14333. Epub 2022 Feb 8. PMID: 35137543; PMCID: PMC8888150
9. Lakshmipriya B., Pottakkat B., Ramkumar G. Deep learning techniques in liver tumour diagnosis using CT and MR imaging – A systematic review. Artif Intell Med. 2023 Jul; 141: 102557. https://doi.org/10.1016/j.artmed.2023.102557. Epub 2023 Apr 29. PMID: 37295904
10. Rich J.M., Bhardwaj L.N., Shah A., et al. Deep learning image segmentation approaches for malignant bone lesions: a systematic review and meta-analysis. Front Radiol. 2023 Aug 8; 3: 1241651. https://doi.org/10.3389/fradi.2023.1241651. PMID: 37614529; PMCID: PMC10442705
11. Черненький И.М., Черненький М.М., Фиев Д.Н., Сирота Е.С. Сегментация почечных структур по изображениям контрастной компьютерной томографии с помощью сверточной нейронной сети. Сеченовский вестник. 2023; 14(1): 39–49. https://doi.org/10.47093/2218-7332.2023.14.1.39-49
12. Shen M.H., Huang C.C., Chen Y.T., et al. Deep learning empowers endoscopic detection and polyps classification: a multiple-hospital study. Diagnostics (Basel). 2023 Apr 19; 13(8): 1473. https://doi.org/10.3390/diagnostics13081473. PMID: 37189575; PMCID: PMC10138002
13. Kuntz S., Krieghoff-Henning E., Kather J.N., et al. Gastrointestinal cancer classification and prognostication from histology using deep learning: Systematic review. Eur J Cancer. 2021 Sep; 155: 200–215. https://doi.org/10.1016/j.ejca.2021.07.012. Epub 2021 Aug 11. PMID: 34391053
14. Ikeda A., Nosato H. Overview of current applications and trends in artificial intelligence for cystoscopy and transurethral resection of bladder tumours. Curr Opin Urol. 2024 Jan 1; 34(1): 27–31. https://doi.org/10.1097/MOU.0000000000001135. Epub 2023 Oct 30. PMID: 37902120
15. Mikołajczyk A., Grochowski M. Data augmentation for improving deep learning in image classification problem. 2018 International Interdisciplinary PhD Workshop (IIPhDW), Świnouście, Poland, 2018. P. 117–122. https://doi.org/10.1109/IIPHDW.2018.8388338
16. Simonyan K., Zisserman A. Very deep convolutional networks for large-scale image recognition. In 3rd International Conference on Learning Representations (ICLR 2015), 1–14. Computational and Biological Learning Society. https://doi.org/10.48550/arXiv.1409.1556
17. Deng J., Dong W., Socher R., et al. ImageNet: A large-scale hierarchical image database. 2009 IEEE Conference on Computer Vision and Pattern Recognition, Miami, FL, USA, 2009. P. 248–255. https://doi.org/10.1109/CVPR.2009.5206848
18. Ronneberger O., Fischer P., Brox T. U-Net: Convolutional networks for biomedical image segmentation. In: Navab N., Hornegger J., Wells W., Frangi A. (eds) Medical Image Computing and Computer-Assisted Intervention – MICCAI 2015. MICCAI 2015. Lecture Notes in Computer Science, vol. 9351. Springer, Cham. https://doi.org/10.1007/978-3-319-24574-4_28
19. Milletari F., Navab N., Ahmadi S.-A. V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation, 2016 Fourth International Conference on 3D Vision (3DV), Stanford, CA, USA, 2016. P. 565–571. https://doi.org/10.1109/3DV.2016.79.
20. Zheng X., He B., Hu Y., et al. Diagnostic accuracy of deep learning and radiomics in lung cancer staging: a systematic review and meta-analysis. Front Public Health. 2022 Jul 18; 10: 938113. https://doi.org/10.3389/fpubh.2022.938113. PMID: 35923964; PMCID: PMC9339706
21. Liu Y., Wu M. Deep learning in precision medicine and focus on glioma. Bioeng Transl Med. 2023 May 31; 8(5): e10553. https://doi.org/10.1002/btm2.10553. PMID: 37693051; PMCID: PMC10486341
22. Рожкова Н.И., Ройтберг П.Г., Варфоломеева А.А. и др. Сегментационная модель скрининга рака молочной железы на основе нейросетевого анализа рентгеновских изображений. Сеченовский вестник. 2020; 11(3): 4–14. https://doi.org/10.47093/2218-7332.2020.11.3.4-14
23. Balkenende L., Teuwen J., Mann R.M. Application of deep learning in breast cancer imaging. Semin Nucl Med. 2022 Sep; 52(5): 584–596. https://doi.org/10.1053/j.semnuclmed.2022.02.003. Epub 2022 Mar 24. PMID: 35339259
24. Wewetzer L., Held L.A., Steinhäuser J. Diagnostic performance of deep-learning-based screening methods for diabetic retinopathy in primary care – A meta-analysis. PLoS One. 2021 Aug 10; 6(8): e0255034. https://doi.org/10.1371/journal.pone.0255034. PMID: 34375355; PMCID: PMC8354436
25. Bechelli S., Delhommelle J. Machine learning and deep learning algorithms for skin cancer classification from dermoscopic images. Bioengineering (Basel). 2022 Feb 27; 9(3): 97. https://doi.org/10.3390/bioengineering9030097. PMID: 35324786; PMCID: PMC8945332
26. Rösler W., Altenbuchinger M., Baeßler B., et al. An overview and a roadmap for artificial intelligence in hematology and oncology. J Cancer Res Clin Oncol. 2023 Aug; 149(10): 7997–8006. https://doi.org/10.1007/s00432-023-04667-5. Epub 2023 Mar 15. PMID: 36920563; PMCID: PMC10374829
27. Shin Y., Qadir H.A., Aabakken L., et al. Automatic colon polyp detection using region based deep CNN and post learning approa ches. IEEE Access. 2018; 6: 40950–40962. https://doi.org/10.1109/ACCESS.2018.2856402
Об авторах
В. С. РозоваАвстралия
Влада Стефановна Розова, PhD, научный сотрудник Школы вычислительных и информационных систем
ул. Граттэн, Парквилл Виктория, г. Мельбурн, 3010
К. Руссо
Австралия
Карло Руссо, PhD, научный сотрудник лаборатории вычислительной нейрохирургии
Балаклава Роуд, Маккуори Парк, г. Сидней, 2109
В. Ю. Лекарев
Россия
Владимир Юрьевич Лекарев, канд. мед. наук, врач-уролог онкологического урологического отделения Университетской клинической больницы № 2
ул. Трубецкая, д. 8, стр. 2, г. Москва, 119048
В. В. Казанцева
Россия
Влада Владимировна Казанцева, студентка 3-го курса
ул. Трубецкая, д. 8, стр. 2, г. Москва, 119048
А. М. Дымов
Россия
Алим Мухамедович Дымов, канд. мед. наук, врач-уролог, старший научный сотрудник НИИ уронефрологии и репродуктивного здоровья человека
ул. Трубецкая, д. 8, стр. 2, г. Москва, 119048
А. С. Ржевский
Россия
Алексей Сергеевич Ржевский, PhD, научный сотрудник лаборатории направленного транспорта лекарственных препаратов Института молекулярной тераностики Научно-технологического парка биомедицины
ул. Трубецкая, д. 8, стр. 2, г. Москва, 119048
А. В. Звягин
Россия
Андрей Васильевич Звягин, д-р физ.-мат. наук, заместитель директора Института молекулярной тераностики Научно-технологического парка биомедицины ФГАОУ ВО «Первый МГМУ им. И.М. Сеченова» Минздрава России (Сеченовский Университет); почетный доцент Школы математических и физических наук Университета Маккуори
Балаклава Роуд, Маккуори Парк, г. Сидней, 2109
ул. Трубецкая, д. 8, стр. 2, г. Москва, 119048